Capítulo 9: Algoritmos de Clustering
¿Qué es el Clustering?
El clustering es una técnica de aprendizaje no supervisado que agrupa datos similares en conjuntos llamados clusters. A diferencia del aprendizaje supervisado, el clustering no requiere datos etiquetados y busca patrones intrínsecos en los datos para identificar grupos naturales.
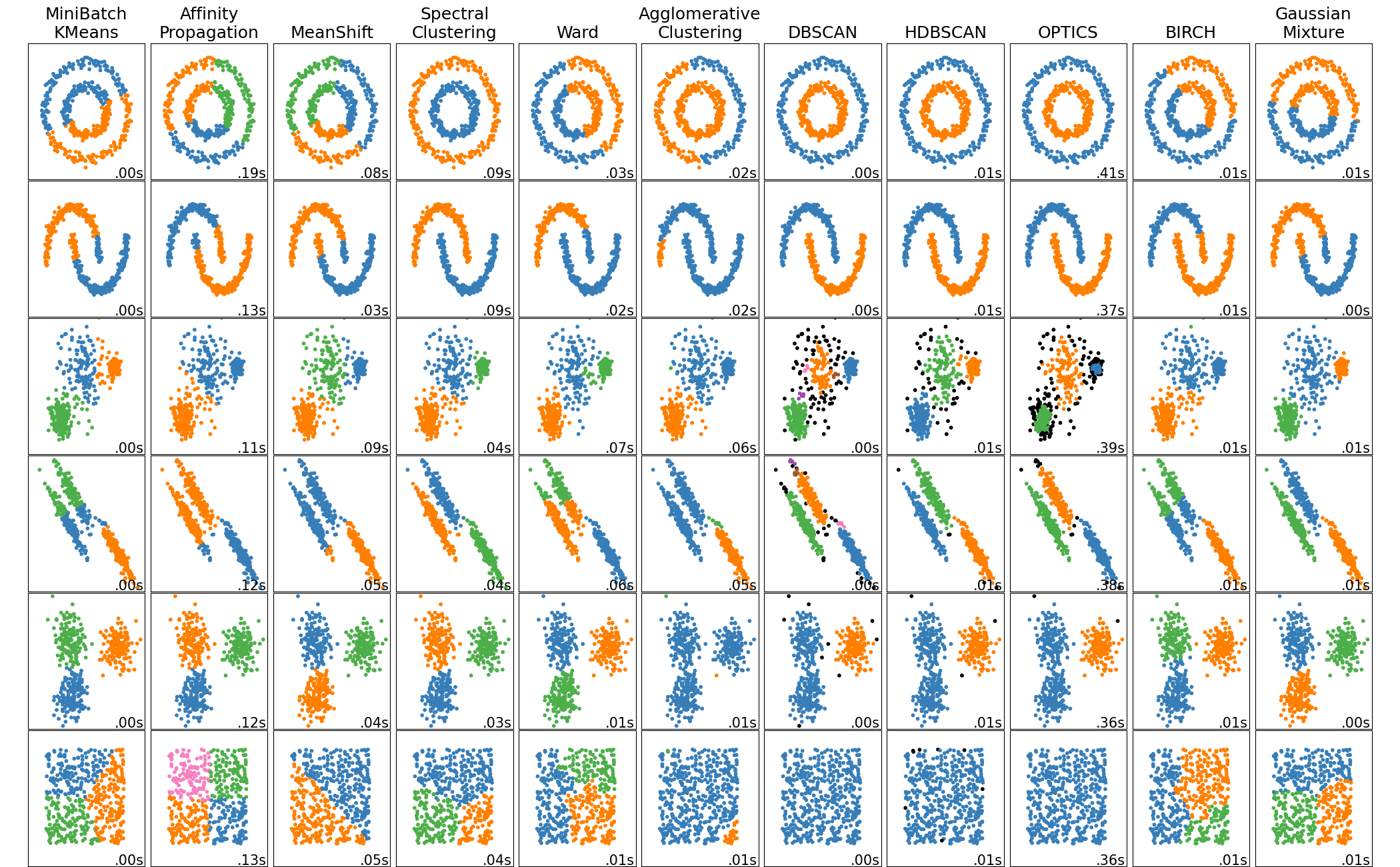
El clustering se utiliza en numerosos campos y aplicaciones:
- Marketing: Segmentación de clientes basada en comportamiento de compra
- Biología: Agrupación de genes con expresión similar
- Astronomía: Clasificación de estrellas y galaxias
- Medicina: Identificación de subtipos de enfermedades
- Análisis de documentos: Organización temática de textos
- Detección de anomalías: Identificación de patrones atípicos
Principales Técnicas de Clustering
Existen varios enfoques para el clustering, cada uno con sus propias fortalezas y características:
Clustering Basado en Centroides
Los algoritmos basados en centroides dividen los datos en K grupos, donde cada grupo está representado por un punto central llamado centroide.
El algoritmo más conocido es K-means, que:
- Asigna cada punto al centroide más cercano
- Recalcula los centroides como el promedio de los puntos asignados
- Repite el proceso hasta la convergencia
Clustering Jerárquico
El clustering jerárquico crea una descomposición anidada de los datos, formando un árbol de clusters (dendrograma).
Existen dos enfoques principales:
- Aglomerativo: Comienza con cada punto como un cluster y los fusiona gradualmente
- Divisivo: Comienza con un solo cluster y lo divide recursivamente
Clustering Basado en Densidad
Los algoritmos basados en densidad identifican regiones densas de puntos separadas por regiones de baja densidad.
El algoritmo más popular es DBSCAN (Density-Based Spatial Clustering of Applications with Noise), que:
- Define clusters como áreas densas conectadas
- Puede detectar clusters de forma arbitraria
- Identifica automáticamente puntos de ruido o valores atípicos
Workflow Completo de Clustering: Análisis del Dataset Iris
A continuación, implementaremos un flujo de trabajo completo de clustering utilizando el famoso dataset Iris. Este ejercicio práctico te permitirá experimentar con diferentes algoritmos de clustering, comparar su rendimiento y optimizar hiperparámetros.
Nuestro objetivo es:
- Implementar algoritmos de clustering (KMeans, Agglomerative, DBSCAN, MeanShift)
- Comparar rendimiento usando métricas de evaluación
- Optimizar hiperparámetros usando PCA para reducción dimensional
1. Importación de Librerías
Comenzamos importando todas las librerías necesarias para nuestro análisis de clustering:
import pandas as pd
import matplotlib.pyplot as plt
import numpy as np
import warnings
warnings.filterwarnings("ignore")
# Librerías de sklearn
from sklearn import metrics
from sklearn.cluster import KMeans, AgglomerativeClustering, DBSCAN, MeanShift
from sklearn import preprocessing
from sklearn.decomposition import PCA
from sklearn.model_selection import ParameterGrid
# Configuración de gráficos
plt.style.use('default')
plt.rcParams['figure.figsize'] = (10, 6)2. Carga y Exploración del Dataset Iris
El dataset Iris es perfecto para practicar clustering ya que contiene 150 muestras de flores con 4 características numéricas y 3 clases naturales (especies). Aunque conocemos las etiquetas verdaderas, las usaremos solo para evaluar qué tan bien nuestros algoritmos de clustering descubren los grupos naturales.
# Cargar el dataset Iris desde sklearn
from sklearn.datasets import load_iris
# Cargar datos
iris_data = load_iris()
iris_df = pd.DataFrame(iris_data.data, columns=iris_data.feature_names)
iris_df['class'] = iris_data.target
# Renombrar columnas para mayor claridad
iris_df.columns = ['sepal-length', 'sepal-width', 'petal-length', 'petal-width', 'class']
print("Forma del dataset:", iris_df.shape)
print("\nPrimeras 5 filas:")
iris_df.head()Forma del dataset: (150, 5)
Primeras 5 filas:
sepal-length sepal-width petal-length petal-width class
0 5.1 3.5 1.4 0.2 0
1 4.9 3.0 1.4 0.2 0
2 4.7 3.2 1.3 0.2 0
3 4.6 3.1 1.5 0.2 0
4 5.0 3.6 1.4 0.2 0Examinemos las características básicas del dataset:
# Información básica del dataset
print("Información del dataset:")
print("\nClases únicas:", iris_df['class'].unique())
print("Distribución de clases:")
print(iris_df['class'].value_counts().sort_index())
print("\nVerificar valores nulos:")
print(iris_df.isnull().sum())
print("\nEstadísticas descriptivas:")
iris_df.describe()Información del dataset:
Clases únicas: [0 1 2]
Distribución de clases:
class
0 50
1 50
2 50
Name: count, dtype: int64
Verificar valores nulos:
sepal-length 0
sepal-width 0
petal-length 0
petal-width 0
class 0
dtype: int643. Preprocesamiento de Datos
Antes de aplicar los algoritmos de clustering, preparamos los datos mezclando el dataset aleatoriamente y separando las características de las etiquetas:
# Mezclar el dataset aleatoriamente
iris_df = iris_df.sample(frac=1, random_state=42).reset_index(drop=True)
print("Dataset después de mezclar:")
print(iris_df.head())
# Separar características y etiquetas
iris_features = iris_df.drop('class', axis=1)
iris_labels = iris_df['class']
print(f"\nForma de características: {iris_features.shape}")
print(f"Forma de etiquetas: {iris_labels.shape}")Dataset después de mezclar:
sepal-length sepal-width petal-length petal-width class
0 6.1 2.8 4.7 1.2 1
1 5.7 3.8 1.7 0.3 0
2 7.7 2.6 6.9 2.3 2
3 6.0 2.9 4.5 1.5 1
4 6.8 2.8 4.8 1.4 1
Forma de características: (150, 4)
Forma de etiquetas: (150,)4. Análisis Exploratorio de Datos
4.1 Análisis Univariable
Comenzamos explorando las distribuciones individuales de cada característica para entender mejor nuestros datos:
# Análisis univariable - Estadísticas por característica
print("Estadísticas descriptivas de las características:")
iris_features.describe()# Visualizar distribuciones
fig, axes = plt.subplots(2, 2, figsize=(8, 6))
axes = axes.ravel()
for i, column in enumerate(iris_features.columns):
axes[i].hist(iris_features[column], bins=20, alpha=0.7, color='skyblue', edgecolor='black')
axes[i].set_title(f'Distribución de {column}')
axes[i].set_xlabel(column)
axes[i].set_ylabel('Frecuencia')
plt.tight_layout()
plt.show()
4.2 Análisis Bivariable
Exploramos las relaciones entre pares de características mediante scatter plots para identificar posibles patrones de agrupamiento:
# Análisis bivariable - Scatter plots
fig, axes = plt.subplots(2, 2, figsize=(8, 6))
# Scatter plot 1: sepal-length vs sepal-width
axes[0,0].scatter(iris_df['sepal-length'], iris_df['sepal-width'], cmap='viridis', s=60, alpha=0.7)
axes[0,0].set_xlabel('Sepal Length')
axes[0,0].set_ylabel('Sepal Width')
axes[0,0].set_title('Sepal Length vs Sepal Width')
# Scatter plot 2: petal-length vs petal-width
axes[0,1].scatter(iris_df['petal-length'], iris_df['petal-width'], cmap='viridis', s=60, alpha=0.7)
axes[0,1].set_xlabel('Petal Length')
axes[0,1].set_ylabel('Petal Width')
axes[0,1].set_title('Petal Length vs Petal Width')
# Scatter plot 3: sepal-length vs petal-length
axes[1,0].scatter(iris_df['sepal-length'], iris_df['petal-length'], cmap='viridis', s=60, alpha=0.7)
axes[1,0].set_xlabel('Sepal Length')
axes[1,0].set_ylabel('Petal Length')
axes[1,0].set_title('Sepal Length vs Petal Length')
# Scatter plot 4: sepal-width vs petal-width
axes[1,1].scatter(iris_df['sepal-width'], iris_df['petal-width'], cmap='viridis', s=60, alpha=0.7)
axes[1,1].set_xlabel('Sepal Width')
axes[1,1].set_ylabel('Petal Width')
axes[1,1].set_title('Sepal Width vs Petal Width')
plt.tight_layout()
plt.show()
Calculamos la matriz de correlación para entender mejor las relaciones lineales entre las variables:
# Matriz de correlación
correlation_matrix = iris_features.corr()
# Visualizar matriz de correlación
plt.figure(figsize=(8, 6))
plt.imshow(correlation_matrix, cmap='coolwarm', aspect='auto')
plt.colorbar()
plt.xticks(range(len(correlation_matrix.columns)), correlation_matrix.columns, rotation=45)
plt.yticks(range(len(correlation_matrix.columns)), correlation_matrix.columns)
plt.title('Matriz de Correlación - Dataset Iris')
# Añadir valores numéricos
for i in range(len(correlation_matrix.columns)):
for j in range(len(correlation_matrix.columns)):
plt.text(j, i, f'{correlation_matrix.iloc[i, j]:.2f}',
ha='center', va='center', color='black')
plt.tight_layout()
plt.show()
5. Implementación de K-Means
Comenzamos con K-Means, uno de los algoritmos de clustering más populares. Aplicamos el algoritmo usando todas las características del dataset y evaluamos su rendimiento:
# Implementar KMeans con todas las características
kmeans_model = KMeans(n_clusters=3, random_state=42, max_iter=1000).fit(iris_features)
# Obtener las etiquetas predichas y centroides
predicted_labels = kmeans_model.labels_
centroids = kmeans_model.cluster_centers_
print("Centroides de los clusters:")
print(pd.DataFrame(centroids, columns=iris_features.columns))
print(f"\nNúmero de puntos por cluster:")
unique, counts = np.unique(predicted_labels, return_counts=True)
for i, count in enumerate(counts):
print(f"Cluster {i}: {count} puntos")Centroides de los clusters:
sepal-length sepal-width petal-length petal-width
0 5.006000 3.428000 1.462000 0.246000
1 5.901613 2.748387 4.393548 1.433871
2 6.850000 3.073684 5.742105 2.071053
Número de puntos por cluster:
Cluster 0: 50 puntos
Cluster 1: 62 puntos
Cluster 2: 38 puntosPara evaluar la calidad del clustering, definimos una función que calcula múltiples métricas de evaluación:
# Función para calcular métricas de evaluación
def evaluate_clustering(true_labels, predicted_labels, data):
"""
Calcula las métricas de evaluación para clustering
"""
silhouette = metrics.silhouette_score(data, predicted_labels)
adjusted_rand = metrics.adjusted_rand_score(true_labels, predicted_labels)
homogeneity = metrics.homogeneity_score(true_labels, predicted_labels)
return {
'Silhouette Score': silhouette,
'Adjusted Rand Score': adjusted_rand,
'Homogeneity Score': homogeneity
}
# Evaluar KMeans
kmeans_metrics = evaluate_clustering(iris_labels, predicted_labels, iris_features)
print("Métricas de evaluación para KMeans:")
print("-" * 40)
for metric, value in kmeans_metrics.items():
print(f"{metric}: {value:.4f}")Métricas de evaluación para KMeans:
----------------------------------------
Silhouette Score: 0.5528
Adjusted Rand Score: 0.7302
Homogeneity Score: 0.7515Visualizamos los resultados comparando el clustering real con el predicho por K-Means:
# Visualizar resultados de KMeans
fig, axes = plt.subplots(1, 2, figsize=(8, 4))
# Gráfico 1: Clustering real vs predicho
axes[0].scatter(iris_features['sepal-length'], iris_features['petal-length'],
c=iris_labels, cmap='viridis', s=40, alpha=0.7, edgecolors='black')
axes[0].set_title('Clustering Real (Ground Truth)')
axes[0].set_xlabel('Sepal Length')
axes[0].set_ylabel('Petal Length')
axes[1].scatter(iris_features['sepal-length'], iris_features['petal-length'],
c=predicted_labels, cmap='viridis', s=40, alpha=0.7, edgecolors='black')
axes[1].scatter(centroids[:, 0], centroids[:, 2], c='red', s=50, marker='X',
edgecolors='black', label='Centroides')
axes[1].set_title('Clustering KMeans')
axes[1].set_xlabel('Sepal Length')
axes[1].set_ylabel('Petal Length')
axes[1].legend()
plt.tight_layout()
plt.show()
6. Comparación de Algoritmos de Clustering
Ahora compararemos el rendimiento de diferentes algoritmos de clustering para determinar cuál funciona mejor con nuestros datos. Implementaremos K-Means, Clustering Aglomerativo, DBSCAN y MeanShift:
# Función para construir y evaluar modelos
def build_and_evaluate_model(clustering_function, data, true_labels, **kwargs):
"""
Construye un modelo de clustering y evalúa su rendimiento
"""
model = clustering_function(**kwargs).fit(data)
metrics_dict = evaluate_clustering(true_labels, model.labels_, data)
return model, metrics_dict
# Diccionario para almacenar resultados
results = {}
models = {}
print("Evaluando algoritmos de clustering...")
print("=" * 60)Evaluando algoritmos de clustering...
============================================================6.1 K-Means
# 1. KMeans
print("1. KMeans")
print("-" * 20)
kmeans_model, kmeans_results = build_and_evaluate_model(
KMeans, iris_features, iris_labels,
n_clusters=3, max_iter=1000
)
results['KMeans'] = kmeans_results
models['KMeans'] = kmeans_model
for metric, value in kmeans_results.items():
print(f"{metric}: {value:.4f}")
print()1. KMeans
--------------------
Silhouette Score: 0.5512
Adjusted Rand Score: 0.7163
Homogeneity Score: 0.73646.2 Clustering Aglomerativo
# 2. Agglomerative Clustering
print("2. Agglomerative Clustering")
print("-" * 30)
agg_model, agg_results = build_and_evaluate_model(
AgglomerativeClustering, iris_features, iris_labels,
n_clusters=3
)
results['Agglomerative'] = agg_results
models['Agglomerative'] = agg_model
for metric, value in agg_results.items():
print(f"{metric}: {value:.4f}")
print()2. Agglomerative Clustering
------------------------------
Silhouette Score: 0.5543
Adjusted Rand Score: 0.7312
Homogeneity Score: 0.76086.3 DBSCAN
# 3. DBSCAN
print("3. DBSCAN")
print("-" * 15)
dbscan_model, dbscan_results = build_and_evaluate_model(
DBSCAN, iris_features, iris_labels,
eps=0.9, min_samples=5
)
results['DBSCAN'] = dbscan_results
models['DBSCAN'] = dbscan_model
for metric, value in dbscan_results.items():
print(f"{metric}: {value:.4f}")
# Información adicional sobre DBSCAN
n_clusters = len(set(dbscan_model.labels_)) - (1 if -1 in dbscan_model.labels_ else 0)
n_noise = list(dbscan_model.labels_).count(-1)
print(f"Número de clusters encontrados: {n_clusters}")
print(f"Puntos de ruido: {n_noise}")
print()3. DBSCAN
---------------
Silhouette Score: 0.6867
Adjusted Rand Score: 0.5681
Homogeneity Score: 0.5794
Número de clusters encontrados: 2
Puntos de ruido: 06.4 MeanShift
# 4. MeanShift
print("4. MeanShift")
print("-" * 15)
from sklearn.cluster import estimate_bandwidth
# Estimar bandwidth automáticamente
bandwidth = estimate_bandwidth(iris_features)
print(f"Bandwidth estimado: {bandwidth:.4f}")
meanshift_model, meanshift_results = build_and_evaluate_model(
MeanShift, iris_features, iris_labels,
bandwidth=bandwidth
)
results['MeanShift'] = meanshift_results
models['MeanShift'] = meanshift_model
for metric, value in meanshift_results.items():
print(f"{metric}: {value:.4f}")
n_clusters_ms = len(set(meanshift_model.labels_))
print(f"Número de clusters encontrados: {n_clusters_ms}")4. MeanShift
---------------
Bandwidth estimado: 1.2021
Silhouette Score: 0.6858
Adjusted Rand Score: 0.5584
Homogeneity Score: 0.5537
Número de clusters encontrados: 26.5 Resumen Comparativo
# Crear tabla comparativa de resultados
results_df = pd.DataFrame(results).T
results_df = results_df.round(4)
print("RESUMEN COMPARATIVO DE ALGORITMOS")
print("=" * 50)
print(results_df)RESUMEN COMPARATIVO DE ALGORITMOS
==================================================
Silhouette Score Adjusted Rand Score Homogeneity Score
KMeans 0.5512 0.7163 0.7364
Agglomerative 0.5543 0.7312 0.7608
DBSCAN 0.6867 0.5681 0.5794
MeanShift 0.6858 0.5584 0.5537# Visualizar comparación
fig, axes = plt.subplots(1, 3, figsize=(14, 5))
metrics_names = list(results_df.columns)
for i, metric in enumerate(metrics_names):
algorithms = results_df.index
values = results_df[metric]
bars = axes[i].bar(algorithms, values, color=['skyblue', 'lightgreen', 'salmon', 'gold'])
axes[i].set_title(f'{metric}')
axes[i].set_ylabel('Score')
axes[i].tick_params(axis='x', rotation=45)
# Añadir valores en las barras
for bar, value in zip(bars, values):
height = bar.get_height()
axes[i].text(bar.get_x() + bar.get_width()/2., height + 0.01,
f'{value:.3f}', ha='center', va='bottom')
plt.tight_layout()
plt.show()
7. Optimización de Hiperparámetros usando PCA
Para mejorar nuestros resultados y facilitar la visualización, aplicamos PCA (Análisis de Componentes Principales) para reducir la dimensionalidad a 2 componentes, manteniendo la mayor parte de la varianza de los datos originales:
# Aplicar PCA para reducir a 2 componentes
pca = PCA(n_components=2, random_state=42)
iris_pca = pca.fit_transform(iris_features)
print("Varianza explicada por cada componente:")
for i, variance in enumerate(pca.explained_variance_ratio_):
print(f"Componente {i+1}: {variance:.4f} ({variance*100:.2f}%)")
print(f"\nVarianza total explicada: {sum(pca.explained_variance_ratio_):.4f} ({sum(pca.explained_variance_ratio_)*100:.2f}%)")
# Crear DataFrame con componentes PCA
iris_pca_df = pd.DataFrame(iris_pca, columns=['PC1', 'PC2'])
iris_pca_df['class'] = iris_labels.values
print(f"\nForma de datos después de PCA: {iris_pca_df.shape}")
iris_pca_df.head()Varianza explicada por cada componente:
Componente 1: 0.9246 (92.46%)
Componente 2: 0.0531 (5.31%)
Varianza total explicada: 0.9777 (97.77%)
Forma de datos después de PCA: (150, 3)# Visualizar datos después de PCA
plt.figure(figsize=(10, 6))
scatter = plt.scatter(iris_pca_df['PC1'], iris_pca_df['PC2'],
c=iris_pca_df['class'], cmap='viridis', s=70, alpha=0.7, edgecolors='black')
plt.xlabel(f'Primera Componente Principal')
plt.ylabel(f'Segunda Componente Principal')
plt.title('Dataset Iris después de PCA (2 componentes)')
plt.grid(True, alpha=0.3)
plt.show()
# Datos para optimización
pca_features = iris_pca_df[['PC1', 'PC2']]
7.1 Optimización de K-Means
Ahora optimizamos los hiperparámetros de cada algoritmo utilizando los datos transformados por PCA:
# Optimización de hiperparámetros para KMeans
print("OPTIMIZACIÓN DE HIPERPARÁMETROS - KMEANS")
print("=" * 50)
# Parámetros a probar
kmeans_params = {'n_clusters': [2, 3, 4, 5]}
parameter_grid = ParameterGrid(kmeans_params)
best_kmeans_score = -1
best_kmeans_params = None
kmeans_results_opt = []
for params in parameter_grid:
model = KMeans(random_state=42, max_iter=1000, **params)
model.fit(pca_features)
silhouette = metrics.silhouette_score(pca_features, model.labels_)
result = {'params': params, 'silhouette_score': silhouette}
kmeans_results_opt.append(result)
print(f"Parámetros: {params} | Silhouette Score: {silhouette:.4f}")
if silhouette > best_kmeans_score:
best_kmeans_score = silhouette
best_kmeans_params = params
print(f"\nMejores parámetros KMeans: {best_kmeans_params}")
print(f"Mejor Silhouette Score: {best_kmeans_score:.4f}")OPTIMIZACIÓN DE HIPERPARÁMETROS - KMEANS
==================================================
Parámetros: {'n_clusters': 2} | Silhouette Score: 0.6810
Parámetros: {'n_clusters': 3} | Silhouette Score: 0.5528
Parámetros: {'n_clusters': 4} | Silhouette Score: 0.4979
Parámetros: {'n_clusters': 5} | Silhouette Score: 0.4258
Mejores parámetros KMeans: {'n_clusters': 2}
Mejor Silhouette Score: 0.68107.2 Optimización de DBSCAN
# Optimización de hiperparámetros para DBSCAN
print("\nOPTIMIZACIÓN DE HIPERPARÁMETROS - DBSCAN")
print("=" * 50)
# Parámetros a probar
dbscan_params = {
'eps': [0.3, 0.5, 0.7, 0.9],
'min_samples': [3, 5, 7]
}
parameter_grid = ParameterGrid(dbscan_params)
best_dbscan_score = -1
best_dbscan_params = None
dbscan_results_opt = []
for params in parameter_grid:
model = DBSCAN(**params)
model.fit(pca_features)
# Verificar que se formaron clusters válidos
n_clusters = len(set(model.labels_)) - (1 if -1 in model.labels_ else 0)
n_noise = list(model.labels_).count(-1)
silhouette = metrics.silhouette_score(pca_features, model.labels_)
result = {'params': params, 'silhouette_score': silhouette,
'n_clusters': n_clusters, 'n_noise': n_noise}
dbscan_results_opt.append(result)
print(f"Parámetros: {params} | Silhouette: {silhouette:.4f} | Clusters: {n_clusters} | Ruido: {n_noise}")
if silhouette > best_dbscan_score:
best_dbscan_score = silhouette
best_dbscan_params = params
print(f"\nMejores parámetros DBSCAN: {best_dbscan_params}")
print(f"Mejor Silhouette Score: {best_dbscan_score:.4f}")OPTIMIZACIÓN DE HIPERPARÁMETROS - DBSCAN
==================================================
Parámetros: {'eps': 0.3, 'min_samples': 3} | Silhouette: 0.6231 | Clusters: 3 | Ruido: 6
Parámetros: {'eps': 0.3, 'min_samples': 5} | Silhouette: 0.6867 | Clusters: 2 | Ruido: 17
Parámetros: {'eps': 0.3, 'min_samples': 7} | Silhouette: 0.6867 | Clusters: 2 | Ruido: 17
Parámetros: {'eps': 0.5, 'min_samples': 3} | Silhouette: 0.5574 | Clusters: 3 | Ruido: 0
Parámetros: {'eps': 0.5, 'min_samples': 5} | Silhouette: 0.5574 | Clusters: 3 | Ruido: 0
Parámetros: {'eps': 0.5, 'min_samples': 7} | Silhouette: 0.5574 | Clusters: 3 | Ruido: 0
Parámetros: {'eps': 0.7, 'min_samples': 3} | Silhouette: 0.6867 | Clusters: 2 | Ruido: 0
Parámetros: {'eps': 0.7, 'min_samples': 5} | Silhouette: 0.6867 | Clusters: 2 | Ruido: 0
Parámetros: {'eps': 0.7, 'min_samples': 7} | Silhouette: 0.6867 | Clusters: 2 | Ruido: 0
Parámetros: {'eps': 0.9, 'min_samples': 3} | Silhouette: 0.6867 | Clusters: 2 | Ruido: 0
Parámetros: {'eps': 0.9, 'min_samples': 5} | Silhouette: 0.6867 | Clusters: 2 | Ruido: 0
Parámetros: {'eps': 0.9, 'min_samples': 7} | Silhouette: 0.6867 | Clusters: 2 | Ruido: 0
Mejores parámetros DBSCAN: {'eps': 0.3, 'min_samples': 5}
Mejor Silhouette Score: 0.68677.3 Optimización de MeanShift
# Optimización de hiperparámetros para MeanShift
print("\nOPTIMIZACIÓN DE HIPERPARÁMETROS - MEANSHIFT")
print("=" * 50)
# Estimar bandwidth base
base_bandwidth = estimate_bandwidth(pca_features)
print(f"Bandwidth base estimado: {base_bandwidth:.4f}")
# Parámetros a probar (múltiplos del bandwidth base)
bandwidth_multipliers = [0.5, 0.8, 1.0, 1.2, 1.5]
meanshift_params = [{'bandwidth': base_bandwidth * mult} for mult in bandwidth_multipliers]
best_meanshift_score = -1
best_meanshift_params = None
meanshift_results_opt = []
for params in meanshift_params:
model = MeanShift(**params)
model.fit(pca_features)
n_clusters = len(set(model.labels_))
silhouette = metrics.silhouette_score(pca_features, model.labels_)
result = {'params': params, 'silhouette_score': silhouette, 'n_clusters': n_clusters}
meanshift_results_opt.append(result)
print(f"Bandwidth: {params['bandwidth']:.4f} | Silhouette: {silhouette:.4f} | Clusters: {n_clusters}")
if silhouette > best_meanshift_score:
best_meanshift_score = silhouette
best_meanshift_params = params
print(f"\nMejores parámetros MeanShift: {best_meanshift_params}")
print(f"Mejor Silhouette Score: {best_meanshift_score:.4f}")OPTIMIZACIÓN DE HIPERPARÁMETROS - MEANSHIFT
==================================================
Bandwidth base estimado: 1.0495
Bandwidth: 0.5248 | Silhouette: 0.2639 | Clusters: 16
Bandwidth: 0.8396 | Silhouette: 0.6858 | Clusters: 2
Bandwidth: 1.0495 | Silhouette: 0.6858 | Clusters: 2
Bandwidth: 1.2594 | Silhouette: 0.5528 | Clusters: 3
Bandwidth: 1.5743 | Silhouette: 0.5528 | Clusters: 3
Mejores parámetros MeanShift: {'bandwidth': 0.8396011904761905}
Mejor Silhouette Score: 0.68588. Resultados Finales y Comparación
Finalmente, entrenamos los modelos con los mejores parámetros encontrados y comparamos los resultados:
# Comparación final con parámetros optimizados
print("\nCOMPARACIÓN FINAL CON PARÁMETROS OPTIMIZADOS")
print("=" * 60)
# Entrenar modelos con mejores parámetros
optimized_results = {}
optimized_models = {}
# KMeans optimizado
if best_kmeans_params:
kmeans_opt = KMeans(random_state=42, max_iter=1000, **best_kmeans_params).fit(pca_features)
kmeans_metrics_opt = evaluate_clustering(iris_labels, kmeans_opt.labels_, pca_features)
optimized_results['KMeans_Optimized'] = kmeans_metrics_opt
optimized_models['KMeans_Optimized'] = kmeans_opt
print(f"KMeans con parámetros: {best_kmeans_params}")
# DBSCAN optimizado (si se encontraron parámetros válidos)
if best_dbscan_params:
dbscan_opt = DBSCAN(**best_dbscan_params).fit(pca_features)
dbscan_metrics_opt = evaluate_clustering(iris_labels, dbscan_opt.labels_, pca_features)
optimized_results['DBSCAN_Optimized'] = dbscan_metrics_opt
optimized_models['DBSCAN_Optimized'] = dbscan_opt
print(f"DBSCAN con parámetros: {best_dbscan_params}")
else:
print("DBSCAN: No se pudieron optimizar parámetros")
# MeanShift optimizado
if best_meanshift_params:
meanshift_opt = MeanShift(**best_meanshift_params).fit(pca_features)
meanshift_metrics_opt = evaluate_clustering(iris_labels, meanshift_opt.labels_, pca_features)
optimized_results['MeanShift_Optimized'] = meanshift_metrics_opt
optimized_models['MeanShift_Optimized'] = meanshift_opt
print(f"MeanShift con parámetros: {best_meanshift_params}")COMPARACIÓN FINAL CON PARÁMETROS OPTIMIZADOS
============================================================
KMeans con parámetros: {'n_clusters': 2}
DBSCAN con parámetros: {'eps': 0.3, 'min_samples': 5}
MeanShift con parámetros: {'bandwidth': 0.8396011904761905}# Mostrar resultados
if optimized_results:
optimized_df = pd.DataFrame(optimized_results).T
optimized_df = optimized_df.round(4)
print("\nRESULTADOS CON PARÁMETROS OPTIMIZADOS:")
print(optimized_df)
else:
print("No se pudieron generar resultados optimizados")RESULTADOS CON PARÁMETROS OPTIMIZADOS:
Silhouette Score Adjusted Rand Score Homogeneity Score
KMeans_Optimized 0.6810 0.5681 0.5794
DBSCAN_Optimized 0.6867 0.5681 0.5794
MeanShift_Optimized 0.6858 0.5681 0.5794# Visualización final de resultados optimizados
n_models = len(optimized_models)
fig, axes = plt.subplots(1, n_models, figsize=(5*n_models, 5))
# Si solo hay un modelo, axes no será una lista
if n_models == 1:
axes = [axes]
colors = ['viridis', 'plasma', 'inferno']
for i, (title, model) in enumerate(optimized_models.items()):
scatter = axes[i].scatter(pca_features['PC1'], pca_features['PC2'],
c=model.labels_, cmap=colors[i % len(colors)],
s=100, alpha=0.7, edgecolors='black')
axes[i].set_title(f'{title}\nSilhouette: {optimized_results[title]["Silhouette Score"]:.4f}')
axes[i].set_xlabel('PC1')
axes[i].set_ylabel('PC2')
axes[i].grid(True, alpha=0.3)
plt.tight_layout()
plt.show()
print("\n¡Análisis de clustering completado!")¡Análisis de clustering completado!
Material de Práctica
Para consolidar los conceptos aprendidos sobre clustering, te invito a practicar con este ejercicio completo:
- Workflow Completo de Clustering - Dataset Iris - Implementa y compara diferentes algoritmos de clustering con optimización de hiperparámetros.
Referencias
Para profundizar en algoritmos de clustering con Python, recomendamos consultar estos recursos:
- Clustering - Scikit-learn Documentation, la documentación oficial de scikit-learn sobre algoritmos de clustering, con ejemplos detallados y comparaciones entre diferentes métodos.
- The 5 Clustering Algorithms Data Scientists Need to Know, un artículo completo de Towards Data Science que explica los algoritmos de clustering más importantes con ejemplos prácticos en Python.